# load packages
library(tidyverse)Si vous êtes présent à la session en direct le lundi 21 octobre, veuillez cliquer ici pour participer à l’enquête.
Tout le matériel nécessaire, y compris le code R et les données, sont disponibles dans le répertoire Github.
Nous utiliserons les packages tidyverse and validate dans ce tutoriel. Vous devrez peut-être les installer si vous ne les avez pas déjà. Pour les installer, exécutez la commande suivante dans votre console R : install.packages("tidyverse", "validate"). Notez que le package tidyverse est volumineux et peut prendre quelques minutes pour s’installer.
Le code de la session en direct est disponible sur Github.
Introduction
Qu’entendons-nous par “data wrangling” (traitement des données)?
Le dictionnaire de Cambridge donne plusieurs significations au verbe wrangle:
to argue with someone about something, especially for a long time (se disputer avec quelqu’un à propos de quelque chose, en particulier pendant une longue période)
to take care of, control, or move animals, especially large animals such as cows or horses (mainly American English) (prendre soin, contrôler ou déplacer des animaux, en particulier les gros animaux comme les vaches ou les chevaux)
to move a person or thing somewhere, usually with difficulty or using force (déplacer une personne ou une chose quelque part, généralement avec difficulté ou en utilisant la force)
to take care of or deal with something, usually when this is difficult (prendre soin de ou gérer quelque chose, généralement lorsque cela est difficile)
Data Wrangling (traitement des données)
Par “traitement des données”, nous entendons ici le processus de vérification et de correction de la qualité et de l’intégrité des données pertinentes pour la modélisation du paludisme, avant toute analyse ultérieure. Ce processus est également connu sous le nom de validation des données.
La validation de données consiste à vérifier divers aspects de votre ensemble de données, tels que les valeurs manquantes, les types de données, les valeurs aberrantes et le respect de règles ou de contraintes spécifiques.
Valider nos données contribue à maintenir leur qualité et leur intégrité, garantissant que toutes les analyses ou décisions prises sur la base des données sont robustes et fiables.
Pourquoi valider les données?
Garantir l’intégrité des données : la validation des données permet d’identifier et de corriger les erreurs, garantissant ainsi l’intégrité de l’ensemble de données.
Améliorer l’exactitude des analyses : des données propres et validées permettent d’obtenir des résultats d’analyse et de modélisation plus précis.
Conformité et normes : la validation des données garantit que les données sont conformes aux règles, normes ou exigences réglementaires prédéfinies.
Prévention des erreurs : la détection précoce des erreurs permet d’éviter les problèmes en aval et de faire gagner du temps lors de leur résolution.
Getting Started
Before you begin, you might want to create a new project in RStudio. This can be done by clicking on the “New Project” button in the upper right corner of the RStudio window. You can then name the project and choose a directory to save it in.
Next, we will load the tidyverse package. This package provides a set of useful functions for data manipulation and visualization. We will use the ggplot2 package to create plots in the later section of this tutorial.
Avant de commencer, il est recommendé de créer un nouveau projet dans RStudio. Pour cela, cliquez sur le bouton “Nouveau projet” dans le coin supérieur droit de la fenêtre RStudio. Vous pouvez ensuite nommer le projet et choisir un répertoire dans lequel l’enregistrer.
Ensuite, nous allons charger le package tidyverse. Ce package fournit un ensemble de fonctions utiles pour la manipulation et la visualisation des données. Nous utiliserons le package ggplot2 pour créer des graphiques dans la dernière section de ce tutoriel.
Ensuite, nous allons télécharger les deux exemples de jeux de données que nous utiliserons dans ce tutoriel. Ceux-ci sont disponibles dans le répertoire GitHub AMMnet Hackathon.
Je suggère de créer un dossier data dans votre projet R, puis nous pourrons télécharger les deux exemples d’ensembles de données afin qu’ils soient enregistrés sur votre ordinateur.
# Create a data folder
dir.create("data")
# Download example data
url <- "https://raw.githubusercontent.com/AMMnet/AMMnet-Hackathon/main/02_data-wrangle/data/"
download.file(paste0(url, "mockdata_cases1.csv"), destfile = "data/mockdata_cases1.csv")
download.file(paste0(url, "mosq_mock1.csv"), destfile = "data/mosq_mock1.csv")
# Load example data
data_cases <- read_csv("data/mockdata_cases1.csv")
mosq_data <- read_csv("data/mosq_mock1.csv")Les deux ensembles de données que nous utiliserons sont mockdata_cases1.csv et mosq_mock1.csv, qui sont des exemples d’ensembles de données fictifs qui devraient être similaires aux données de surveillance des cas de paludisme et de collecte de moustiques sur le terrain, respectivement. Dans les sections suivantes, nous utiliserons mockdata_cases1.csv et mosq_mock1.csv pour introduire les concepts de nettoyage et de caractérisation des données dans R.
1. Vérifier les données pour détecter d’éventuelles erreurs
La prévalence est une fraction définie dans [0,1]
Note: Une prévalence de 0 ou 1, bien que non statistiquement erronée, doit être vérifiée pour en garantir l’exactitude.
Quelles observations comportent des erreurs ?
# Erroneous values for prevalence
data_cases%>%
dplyr::filter(prev <= 0 | prev >= 1)# A tibble: 3 × 10
location month year ages total positive xcoord ycoord prev time_order_loc
<chr> <dbl> <dbl> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 mordor 4 2018 15_a… 91 23 -20.0 30.5 25.3 4
2 neverwh… 2 2019 15_a… 22 -1 -20.8 29.6 -0.0455 14
3 neverwh… 3 2018 unde… 25 0 -19.8 30.2 0 3Commentaire: Nous avons deux observations avec des valeurs non-logiques de prev : 25.3and -0.455, et une ligne avec prev égale zéro pour un certain mois.
Programmation défensive
Remarque: L’utilisation de “::” nous permet d’appeler une fonction à partir d’un package spécifique de R. Il arrive que si le package de base “stats” est appelé en premier, la fonction de filter, si elle n’est pas spécifiée avec le package R, échoue.
# Erroneous values for prevalence
data_cases%>%
stats::filter(prev < 0 | prev > 1) Nous corrigeons les deux prévalences en recalculant
Une bonne pratique est de laisser les données d’origine intactes (avantage de R sur Stata):
# Update erroneous values for prevalence
data_prev <- data_cases%>%
dplyr::mutate(prev_updated=positive/total)Nous avons une observation avec une valeur négative par erreur.
Quelles sont vos options?
Ne jamais supprimer les données
Interroger et demander à l’équipe de gestion des données de procéder aux investigations nécessaires et de procéder à une correction.
data_prev%>%
dplyr::filter(prev_updated <= 0 | prev_updated >= 1)# A tibble: 2 × 11
location month year ages total positive xcoord ycoord prev time_order_loc
<chr> <dbl> <dbl> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 neverwh… 2 2019 15_a… 22 -1 -20.8 29.6 -0.0455 14
2 neverwh… 3 2018 unde… 25 0 -19.8 30.2 0 3
# ℹ 1 more variable: prev_updated <dbl>Pour l’instant (afin de poursuivre cette démonstration), nous abandonnons les observations problématiques.
Pourquoi cela ne fonctionne-t-il pas ?
# Filter erroneous values for prevalence, wrong way
data_use <- data_prev%>%
dplyr::filter (prev_updated >= 0 | prev_updated <= 1)Pourquoi cela ne fonctionne-t-il pas ?
# Filter erroneous values for prevalence
data_use <- data_prev%>%
dplyr::filter (prev_updated >= 0 )%>%
dplyr::filter (prev_updated <= 1)
data_use%>%
dplyr::filter(prev_updated <= 0 | prev_updated >= 1)# A tibble: 1 × 11
location month year ages total positive xcoord ycoord prev time_order_loc
<chr> <dbl> <dbl> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 neverwhere 3 2018 unde… 25 0 -19.8 30.2 0 3
# ℹ 1 more variable: prev_updated <dbl>Schemas
Pour éviter que des erreurs n’apparaissent dans vos données, vous devez définir un schéma qui accompagne vos données enregistrées. Un schéma est un document qui énonce les règles relatives aux types de données et aux valeurs ou plages attendues dans une colonne particulière de votre bloc de données.
Par exemple, pour la prévalence, nous savons qu’il doit s’agir d’un nombre réel compris entre zéro et un.
Le package R validate peut être utilisé pour créer un schéma pour votre trame de données:
# Filter erroneous values for prevalence
library(validate)
schema <- validate::validator(prev >= 0,
prev <= 1,
positive >= 0)
out <- validate::confront(data_cases, schema)
summary(out) name items passes fails nNA error warning expression
1 V1 514 513 1 0 FALSE FALSE prev - 0 >= -1e-08
2 V2 514 513 1 0 FALSE FALSE prev - 1 <= 1e-08
3 V3 514 513 1 0 FALSE FALSE positive - 0 >= -1e-08En utilisant le schéma des colonnes prev et positive, nous aurions pu facilement détecter les trois entrées problématiques. Pour plus de détails, vous pouvez consulter la vignette du package validate.
Remarque: La prochaine fois que vous recevrez des données de vos collaborateurs, n’oubliez pas de leur demander le fichier de schéma associé (par exemple, au format YAML). Bonne chance !
2. Consultez les statistiques récapitulatives
Statistiques agrégées par lieu de collecte (pour toutes les dates)
# Summary statistics
data_use%>%
dplyr::group_by(location)%>%
dplyr::summarise(nobs=n(),
mean_prev=mean(prev_updated),
min_prev=min(prev_updated),
max_prev=max(prev_updated))# A tibble: 5 × 5
location nobs mean_prev min_prev max_prev
<chr> <int> <dbl> <dbl> <dbl>
1 mordor 105 0.314 0.158 0.488
2 narnia 104 0.326 0.08 0.488
3 neverwhere 95 0.301 0 0.486
4 oz 104 0.255 0.0714 0.459
5 wonderland 105 0.382 0.194 0.535Statistiques agrégées par lieu de collecte et par année
Le tableau s’allonge. Il pourrait être trop compliqué d’ajouter des contrôles par mois et par groupe d’âge.
Remarque: Pourquoi n’y a-t-il que 3 mesures en 2020?
# Summary statistics by location
data_use%>%
dplyr::group_by(location, year)%>%
dplyr::summarise(nobs=n(),
mean_prev=mean(prev_updated),
min_prev=min(prev_updated),
max_prev=max(prev_updated))# A tibble: 15 × 6
# Groups: location [5]
location year nobs mean_prev min_prev max_prev
<chr> <dbl> <int> <dbl> <dbl> <dbl>
1 mordor 2018 36 0.318 0.206 0.473
2 mordor 2019 36 0.313 0.170 0.451
3 mordor 2020 33 0.312 0.158 0.488
4 narnia 2018 36 0.340 0.138 0.449
5 narnia 2019 36 0.361 0.216 0.488
6 narnia 2020 32 0.270 0.08 0.483
7 neverwhere 2018 36 0.304 0 0.45
8 neverwhere 2019 56 0.298 0.0370 0.486
9 neverwhere 2020 3 0.307 0.04 0.473
10 oz 2018 35 0.252 0.0714 0.459
11 oz 2019 36 0.254 0.0861 0.446
12 oz 2020 33 0.260 0.112 0.405
13 wonderland 2018 36 0.365 0.255 0.454
14 wonderland 2019 36 0.388 0.194 0.535
15 wonderland 2020 33 0.393 0.276 0.476data_prev et data_use
- Créez un tableau indiquant le nombre d’entrées de données par groupe d’âge et par lieu de collecte pour chacun d’eux!
- Dans quel groupe d’âge et dans quel localité les observations ont-elles été supprimées?
Un peu plus avancé. Utilisation de listes (ce n’est pas le sujet du cours mais c’est un point important).
# Summary statistics by location
data_use_list <- data_use%>%
dplyr::group_split(location)Ou utilisez la librairie purrr :
# Summary statistics by location, map summary function
library(purrr)
data_use_age_summary <- purrr::map(.x=seq(length(data_use_list)),
.f=function(x){
data_use_list[[x]]%>%
dplyr::group_by(location,year,ages)%>%
dplyr::summarise(nobs=n(),
mean_prev=mean(prev_updated),
min_prev=min(prev_updated),
max_prev=max(prev_updated))
})Concentrons-nous maintenant sur le premier objet de la liste (mordor)
Nous savons que les femmes enceintes et les enfants de moins de 5 ans sont les plus vulnérables.
Output (ages) n’est pas ordonné comme nous le souhaiterions (chronologiquement).
# Summary statistics by location
data_mordor <- data_use_age_summary[[1]]
data_mordor# A tibble: 9 × 7
# Groups: location, year [3]
location year ages nobs mean_prev min_prev max_prev
<chr> <dbl> <chr> <int> <dbl> <dbl> <dbl>
1 mordor 2018 15_above 12 0.270 0.206 0.369
2 mordor 2018 5_to_14 12 0.335 0.219 0.427
3 mordor 2018 under_5 12 0.348 0.259 0.473
4 mordor 2019 15_above 12 0.266 0.170 0.377
5 mordor 2019 5_to_14 12 0.278 0.176 0.390
6 mordor 2019 under_5 12 0.394 0.315 0.451
7 mordor 2020 15_above 11 0.255 0.158 0.333
8 mordor 2020 5_to_14 11 0.352 0.258 0.488
9 mordor 2020 under_5 11 0.330 0.190 0.4223. Utilisation des graphiques
Nous devons évaluer l’évolution de la prévalence pour toutes les régions par mois
#Plotting evolution over time
evolution_plot <- ggplot2::ggplot(data=data_use_ordered,
mapping=aes(x=month,
y=prev_updated,
group=location,
colour=location))+
ggplot2::geom_line(lwd=1.1)+
ggplot2::facet_wrap(~year)+
ggplot2::theme_bw()+
ggplot2::xlab("Month of the Year")+
ggplot2::ylab("Prevalence")+
ggplot2::scale_x_discrete(limits=factor(1:12),
labels=c("J","F","M",
"A","M","J",
"J","A","S",
"O","N","D"))+
ggplot2::scale_y_continuous(breaks=seq(from=0,
to=0.7,
by=0.1))
evolution_plot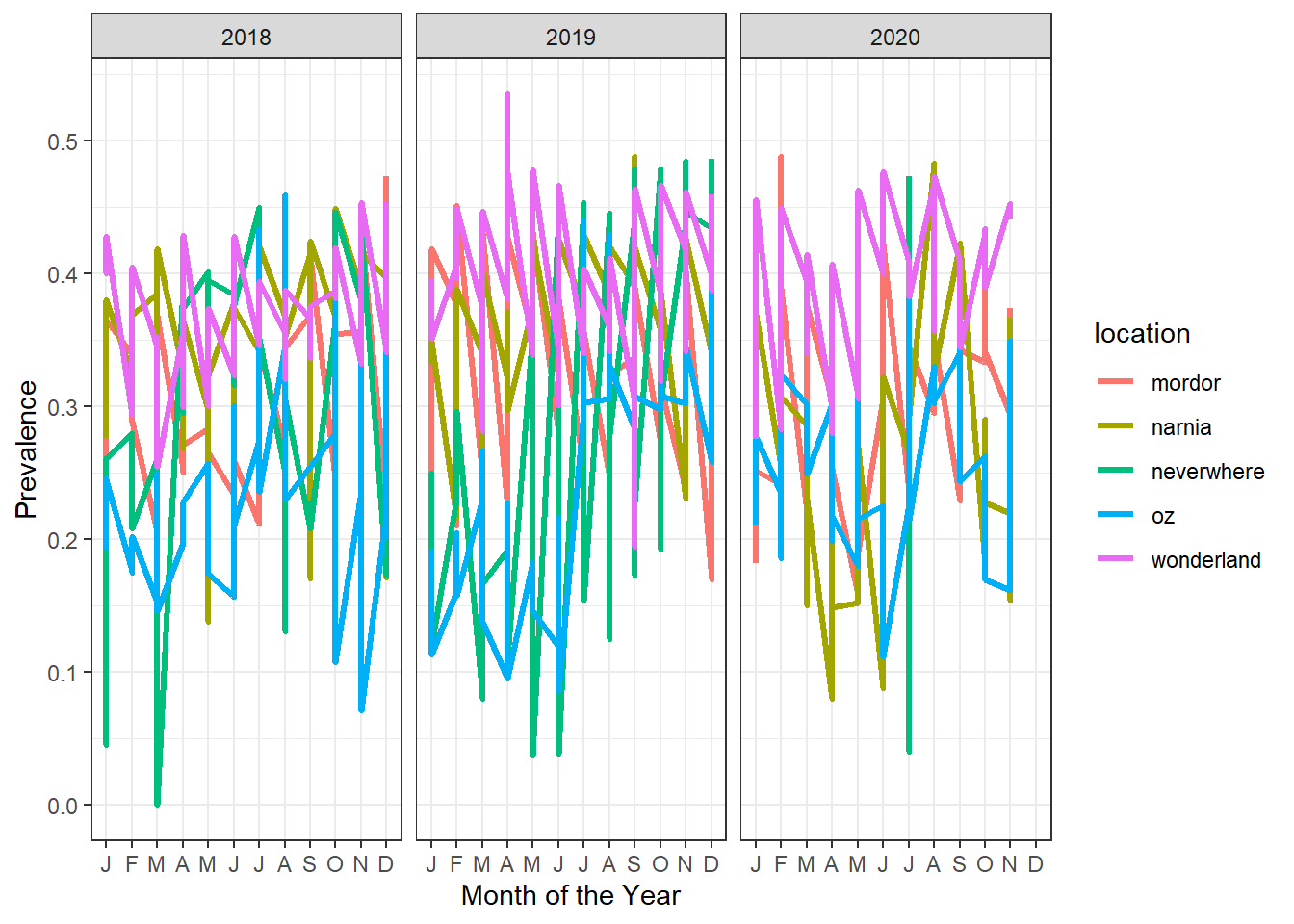
Observation: Graphique de prévalence avec des lignes verticales par mois et par an: nous avons plusieurs sous-groupes pour les données de prévalence et nous traçons des facettes pour les différentes catégories de age_group.
#Plotting evolution over time, fix 1
evolution_plot_ages <- ggplot2::ggplot(data=data_use_ordered,
mapping=aes(x=month,
y=prev_updated,
group=location,
colour=location))+
ggplot2::geom_line(lwd=1.1)+
ggplot2::facet_wrap(age_group~year)+
ggplot2::theme_bw()+
ggplot2::xlab("Month of the Year")+
ggplot2::ylab("Prevalence")+
ggplot2::scale_x_discrete(limits=factor(1:12),
labels=c("J","F","M",
"A","M","J",
"J","A","S",
"O","N","D"))+
ggplot2::scale_y_continuous(breaks=seq(from=0,
to=0.7,
by=0.1))
evolution_plot_ages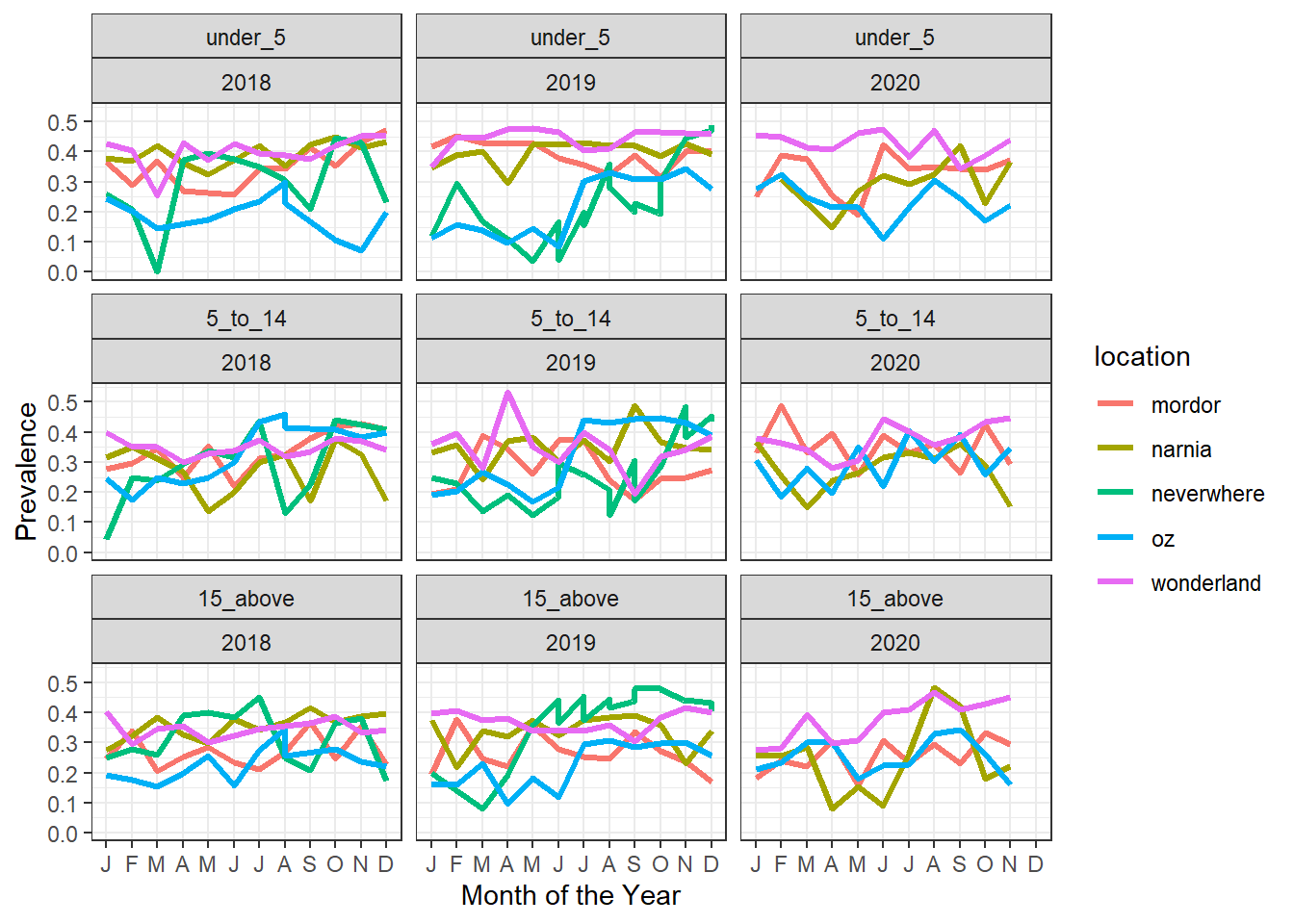
Observation : QIl y a quelques améliorations, mais nous avons toujours des lignes verticales, peut-être avons-nous d’autres variables de groupe. Regardons uniquement les lignes qui ont plus d’une entrée par emplacement, mois, année, groupe d’âge.
#Plotting evolution over time, fix 2
data_use_ordered%>%
group_by(location,month,year,age_group)%>%
tally()%>%
filter(n>1)%>%
left_join(data_use_ordered)# A tibble: 48 × 13
# Groups: location, month, year [8]
location month year age_group n ages total positive xcoord ycoord
<chr> <dbl> <dbl> <fct> <int> <chr> <dbl> <dbl> <dbl> <dbl>
1 neverwhere 6 2019 under_5 2 under_5 24 4 -20.6 30.7
2 neverwhere 6 2019 under_5 2 under_5 26 1 -20.5 30.7
3 neverwhere 6 2019 5_to_14 2 5_to_14 27 5 -19.7 30.0
4 neverwhere 6 2019 5_to_14 2 5_to_14 27 8 -19.3 30.2
5 neverwhere 6 2019 15_above 2 15_above 70 31 -19.4 29.4
6 neverwhere 6 2019 15_above 2 15_above 74 27 -19.2 29.2
7 neverwhere 7 2019 under_5 2 under_5 25 5 -20.0 29.1
8 neverwhere 7 2019 under_5 2 under_5 26 4 -20.7 28.6
9 neverwhere 7 2019 5_to_14 2 5_to_14 27 7 -18.8 29.3
10 neverwhere 7 2019 5_to_14 2 5_to_14 23 6 -20.4 29.8
# ℹ 38 more rows
# ℹ 3 more variables: prev <dbl>, time_order_loc <dbl>, prev_updated <dbl>Observation: OK, nous voyons qu’au sein d’une même localité, il existe plusieurs points de données de prévalence même si les coordonnées xcoord et ycoord diffèrent. Afin d’avoir un seul graphique par lieu, nous pourrions faire la moyenne de xcoord et ycoord dans chaque localité. Il peut aussi s’agir d’enregistrements dupliqués, puisque xcoord et ycoord sont très proches?
#Plotting evolution over time, fix 3
data_use_ordered%>%
group_by(location,month,year,age_group)%>%
summarize(prev_updated_mean=mean(prev_updated),
prev_updated_min=min(prev_updated),
prev_updated_max=max(prev_updated))%>%
ggplot2::ggplot(mapping=aes(x=month,
y=prev_updated_mean,
file=location,
group=location,
colour=location))+
ggplot2::geom_line(lwd=1.1)+
ggplot2::facet_wrap(age_group~year)+
ggplot2::theme_bw()+
ggplot2::xlab("Month of the Year")+
ggplot2::ylab("Prevalence")+
ggplot2::scale_x_discrete(limits=factor(1:12),
labels=c("J","F","M",
"A","M","J",
"J","A","S",
"O","N","D"))+
ggplot2::scale_y_continuous(breaks=seq(from=0,
to=0.7,
by=0.1))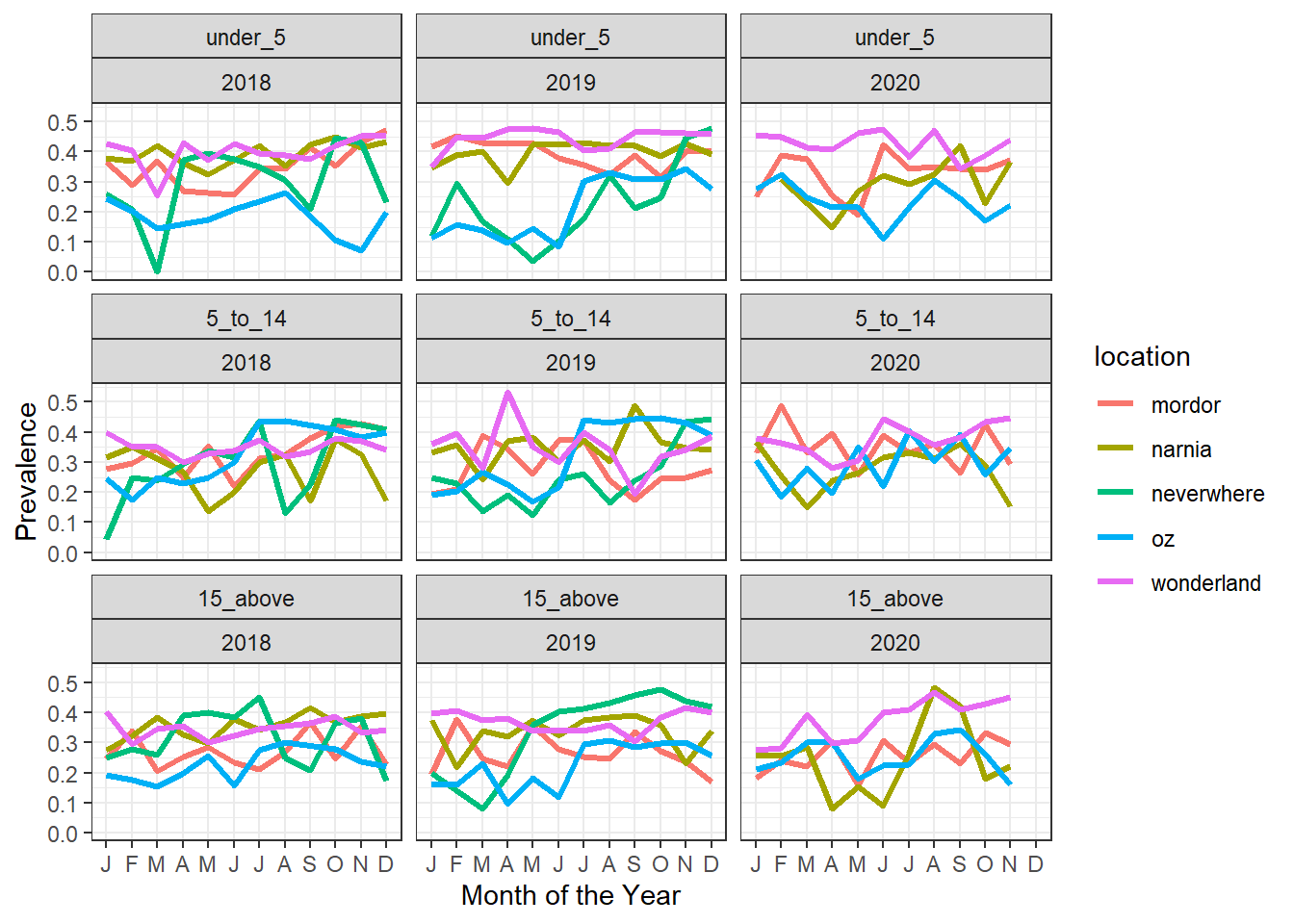
Observation: La prévalence est très variable tout au long de l’année selon les endroits en moyenne; Wonderland étant affecté par une prévalence élevée tandis qu’Oz a la prévalence la plus faible.
Il faut vérifier (pas seulement la prévalence) mais aussi le nombre de cas et le nombre total de personnes vulnérables
#Check case count
data_use_ordered_long <- tidyr::pivot_longer(data=data_use_ordered,
cols=c("positive","total"),
names_to="Outcome",
values_to="counts")
mordor_stacked_bar_graph <- ggplot2::ggplot(data=data_use_ordered_long%>%
dplyr::filter(location=="mordor"),
mapping=aes(x=month,
y=counts,
fill=Outcome))+
ggplot2::scale_x_discrete(limits=factor(1:12),
labels=c("J","F","M",
"A","M","J",
"J","A","S",
"O","N","D"))+
ggplot2::geom_bar(position="stack", stat="identity")+
ggplot2::facet_wrap(~year)+
ggplot2::theme_bw()+
ggplot2::xlab("Month of the Year")+
ggplot2::ylab("Count")
mordor_stacked_bar_graph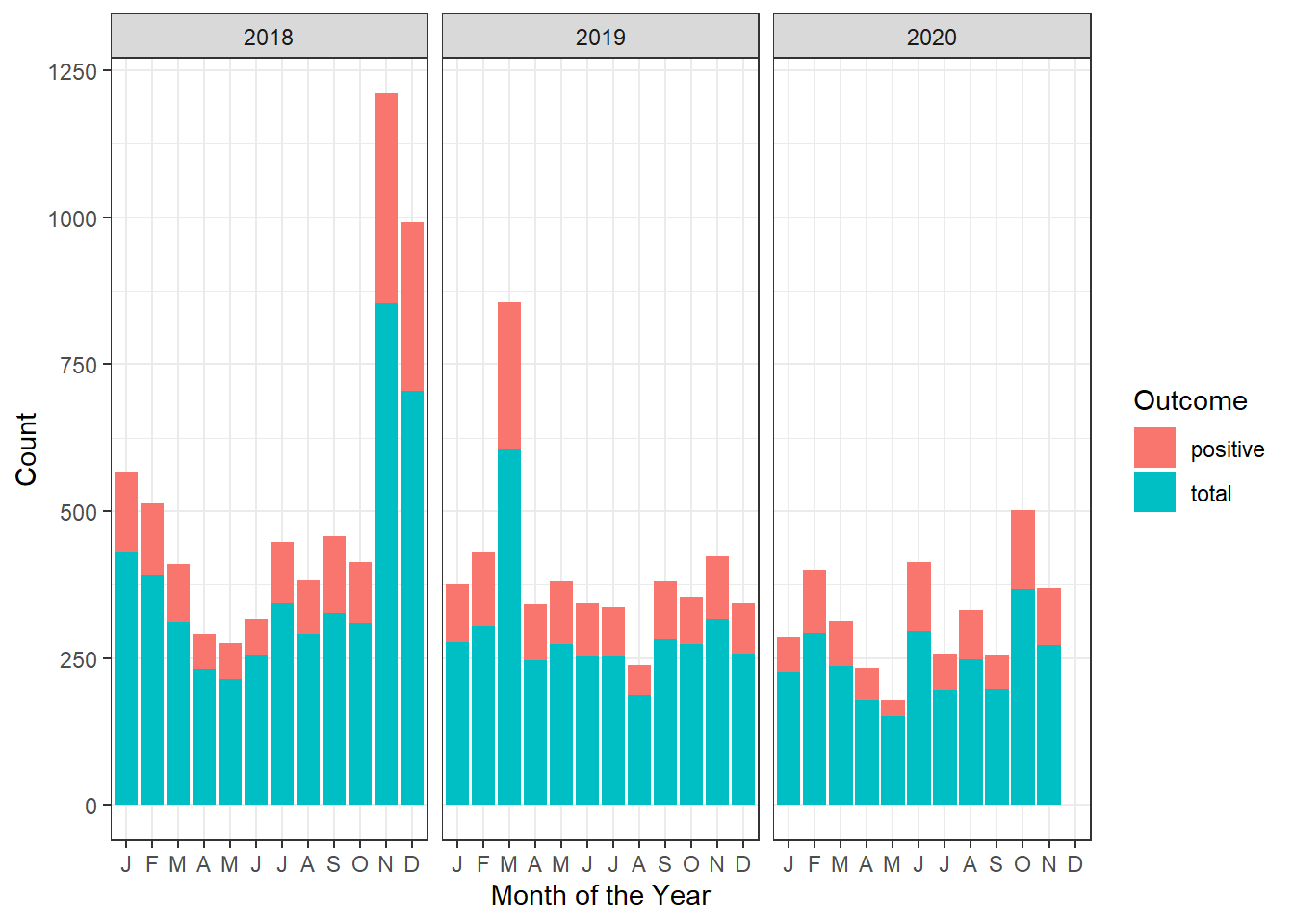
Observation: Le graphique à barres empilées additionne les nombres de positifs et les totaux. Il est préférable de les afficher côte à côte car les nombres de positifs sont un sous-ensemble des nombres totaux. Ceci est spécifié par l’argument position="dodge" dans la géométrie geom_bar de ggplot2.
#Case count, bargraph dodge
mordor_dodged_bar_graph <- ggplot2::ggplot(data=data_use_ordered_long%>%
dplyr::filter(location=="mordor"),
mapping=aes(x=month,
y=counts,
fill=Outcome))+
ggplot2::scale_x_discrete(limits=factor(1:12),
labels=c("J","F","M",
"A","M","J",
"J","A","S",
"O","N","D"))+
ggplot2::geom_bar(position="dodge", stat="identity")+
ggplot2::facet_wrap(~year)+
ggplot2::theme_bw()+
ggplot2::xlab("Month of the Year")+
ggplot2::ylab("Count")
mordor_dodged_bar_graph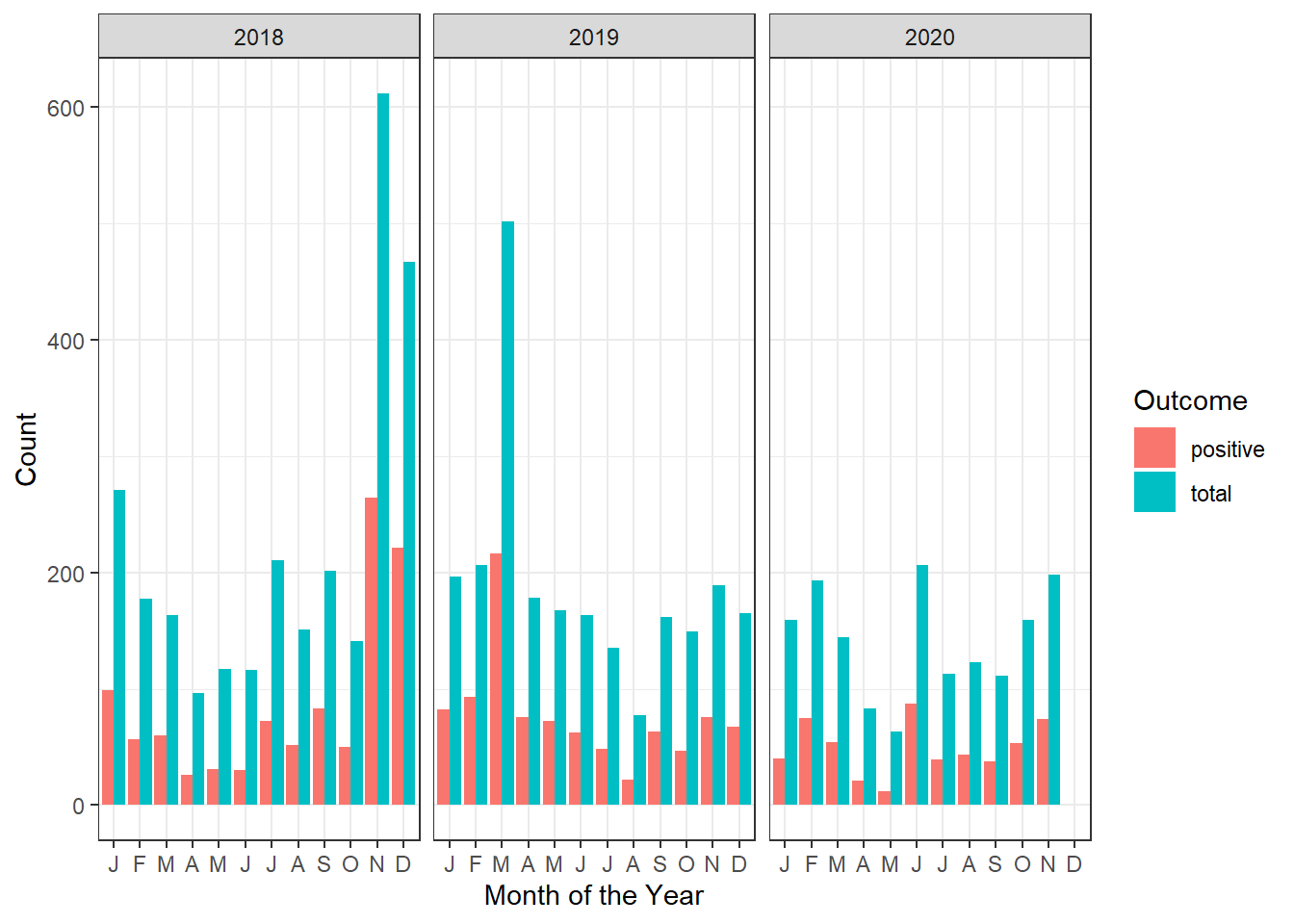
L’ensemble de données sur les moustiques
Jetons un oeil à l’ensemble de données mosq_data.
Nous vérifions la cohérence de cet ensemble de données en affichant un tableau des valeurs enregistrées par colonne:
mosq_data %>%
map( function(x) table(x) )$session
x
1 2
52 52
$Village
x
naernia narnia
2 102
$Compound.ID
x
1 2 3 4
26 26 26 26
$Method
x
ALC HLC
1 103
$Location
x
Indoor Outdoor
52 52
$hour
x
01h-02h 02h-03h 03h-04h 04h-05h 05h-06h 06h-07h 07h-08h 19h-20h 20h-21h 21h-22h
8 8 8 8 8 8 8 8 8 8
22h-23h 23h-24h 24h-01h
8 8 8
$ag.Male
x
0 3 4 5 6 7 14 16 20 22 27 35
93 1 1 1 1 1 1 1 1 1 1 1
$Ag.unfed
x
0 1 2 3 4 5 6 7 8 10 20
57 13 7 8 4 4 2 4 2 1 2
$Ag.halffed
x
0 3 4 5 8 9
92 3 3 3 1 2
$Ag.fed
x
0 1 3 5
88 7 3 6
$Ag.grsgr
x
0 1 2 3 4 6 8 12 17 20 23 27 35 37
70 13 6 1 2 1 3 2 1 1 1 1 1 1
$tot.gamb
x
0 1 2 3 4 5 6 7 8 9 10 11 12 13 14 23
40 10 10 9 8 6 3 2 1 1 5 2 1 2 2 2
$Culex.male
x
0
104
$Culex.female
x
0 1 2
94 9 1
$Mansonia.male
x
0 1
103 1
$Mansonia.female
x
0 1 2
90 11 3
$Aedes.male
x
0
104
$Aedes.female
x
0 1 2
98 5 1 Il semblerait que nous ayons quelques fautes de frappe dans les noms de Method and Village.
- Créez un schéma qui fournit des règles pour les strings (c’est-à-dire les chaînes de caractères) attendues dans les colonnes
MethodetVillage. - Utiliser la syntaxe fournie ici
schema <- validate::validator(Method%in%c("HLC"),
Village%in%c("narnia"))
out <- validate::confront(mosq_data, schema)
summary(out) name items passes fails nNA error warning expression
1 V1 104 103 1 0 FALSE FALSE Method %vin% c("HLC")
2 V2 104 102 2 0 FALSE FALSE Village %vin% c("narnia")Les colonnes Village et Method semblent avoir des erreurs de saisie de données. Nous devons corriger cela.
mosq_data<-mosq_data%>%
mutate(Method=ifelse(Method=="ALC","HLC",Method),
Village=ifelse(Village=="naernia","narnia",Village))Il semble que les différentes colonnes concernent les tailles de population d’Anopheles Gambiae. Modifions les noms des colonnes en utilisant rename du package tidyverse.
mosq_data%>%
rename("AnophelesGambiae.male"="ag.Male",
"AnophelesGambiae.unfed"="Ag.unfed",
"AnophelesGambiae.halffed"="Ag.halffed",
"AnophelesGambiae.fed"="Ag.fed",
"AnophelesGambiae.gravid"="Ag.grsgr")->mosq_dataIl semble que tot.gamb devrait compter le nombre total d’Anopheles Gambiae. Vérifions:
mosq_data%>%
mutate(AnophelesGambiae_total=AnophelesGambiae.male+AnophelesGambiae.unfed+AnophelesGambiae.halffed+AnophelesGambiae.fed+AnophelesGambiae.gravid)->mosq_data
mosq_data%>%
filter(AnophelesGambiae_total!=tot.gamb)%>%select(AnophelesGambiae_total,tot.gamb)# A tibble: 11 × 2
AnophelesGambiae_total tot.gamb
<dbl> <dbl>
1 12 0
2 16 2
3 0 6
4 24 8
5 24 1
6 74 12
7 54 3
8 70 1
9 34 2
10 40 2
11 46 0Donc 11 lignes sur 104 présentent cette divergence. Gardons plutôt Anopheles.total, puisqu’il a été calculé à partir des données.
Comme le statut des Anopheles est mutuellement exclusif dans les données HLC, nous pouvons dessiner un graphique à barres empilées, avec la couleur des barres définie par le statut. Pour produire un tel graphique efficacement dans ggplot2, nous devons faire pivoter le tableau.
Ici en particulier, nous voulons passer d’un format large à un format long afin d’obtenir une colonne décrivant le statut des moustiques Anopheles. Nous utiliserons notamment l’argument names_sep de la fonction pivot_longer pour séparer par exemple le nom de colonne AnophelesGambiae.male et utiliser male comme niveau dans une nouvelle colonne appelée status. Il en va de même pour les autres noms de colonnes.
Intégrer les variables session, Village, Compound.ID, Method, Location, hour, AnophelesGambiae_total dans la définition de la variable de regroupement aidera à conserver ces variables dans le tableau au format long.
mosq_data%>%
group_by(session,Village,Compound.ID,Method,Location,hour,AnophelesGambiae_total)%>%
select(contains("AnophelesGambiae."))%>%
pivot_longer(cols=contains("AnophelesGambiae."),names_sep="AnophelesGambiae.",names_to=c(NA,"status"),values_to = "AnophelesGambiae")->mosq_data_gamb_wide
mosq_data_gamb_wide%>%
ggplot()+
geom_bar(aes(x=hour,y=AnophelesGambiae,fill=status),position="stack",stat="identity")+
scale_x_discrete(guide = guide_axis(angle = 60))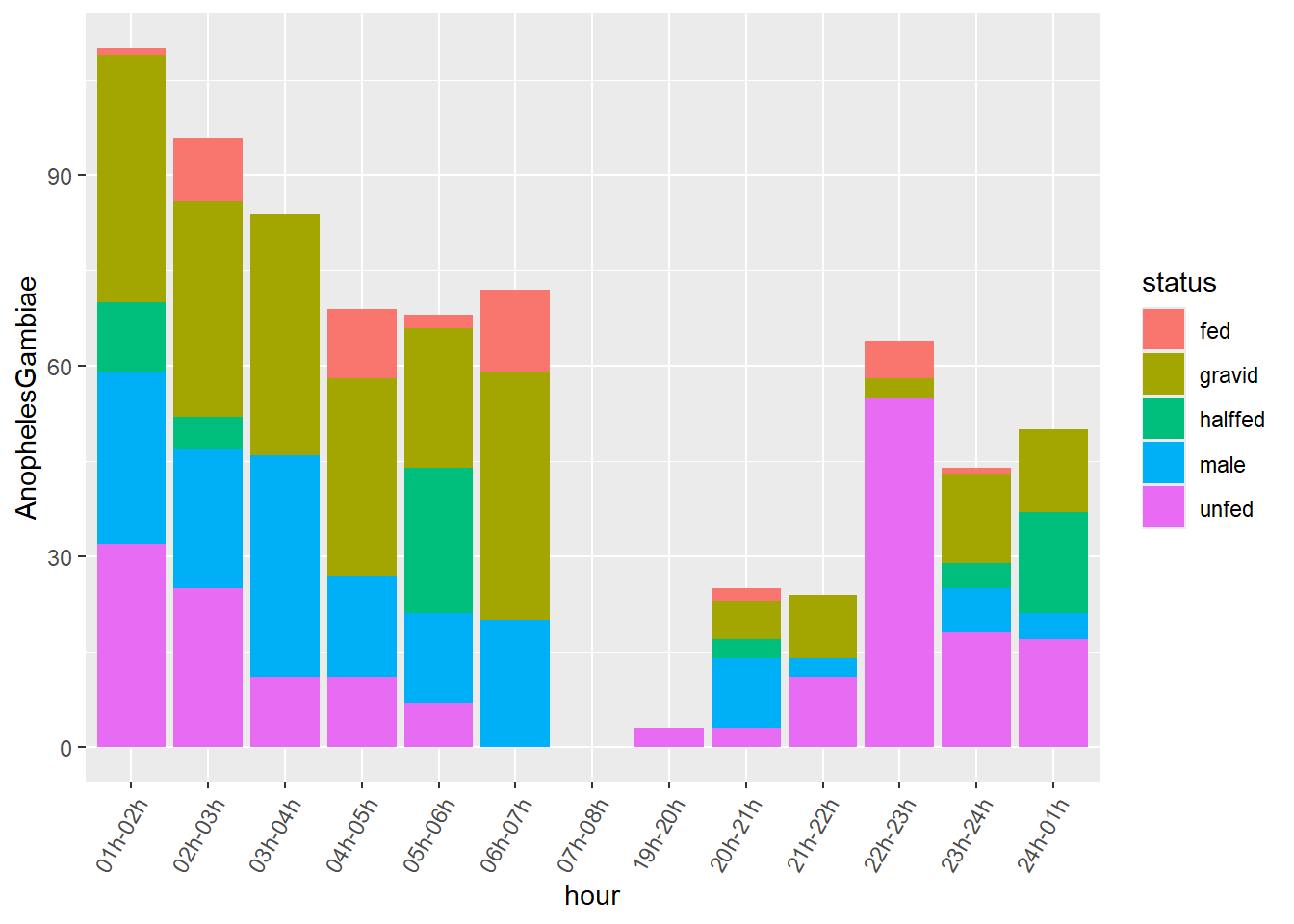
Observation: Nous avons plusieurs valeurs pour Compound.ID. La géométrie geom_bar les additionne automatiquement dans le graphique. Nous pouvons utiliser facet_wrap pour voir ces strates:
mosq_data_gamb_wide%>%
ggplot()+
geom_bar(aes(x=hour,y=AnophelesGambiae,fill=status),position="stack",stat="identity")+
scale_x_discrete(guide = guide_axis(angle = 60))+
facet_wrap(~Compound.ID)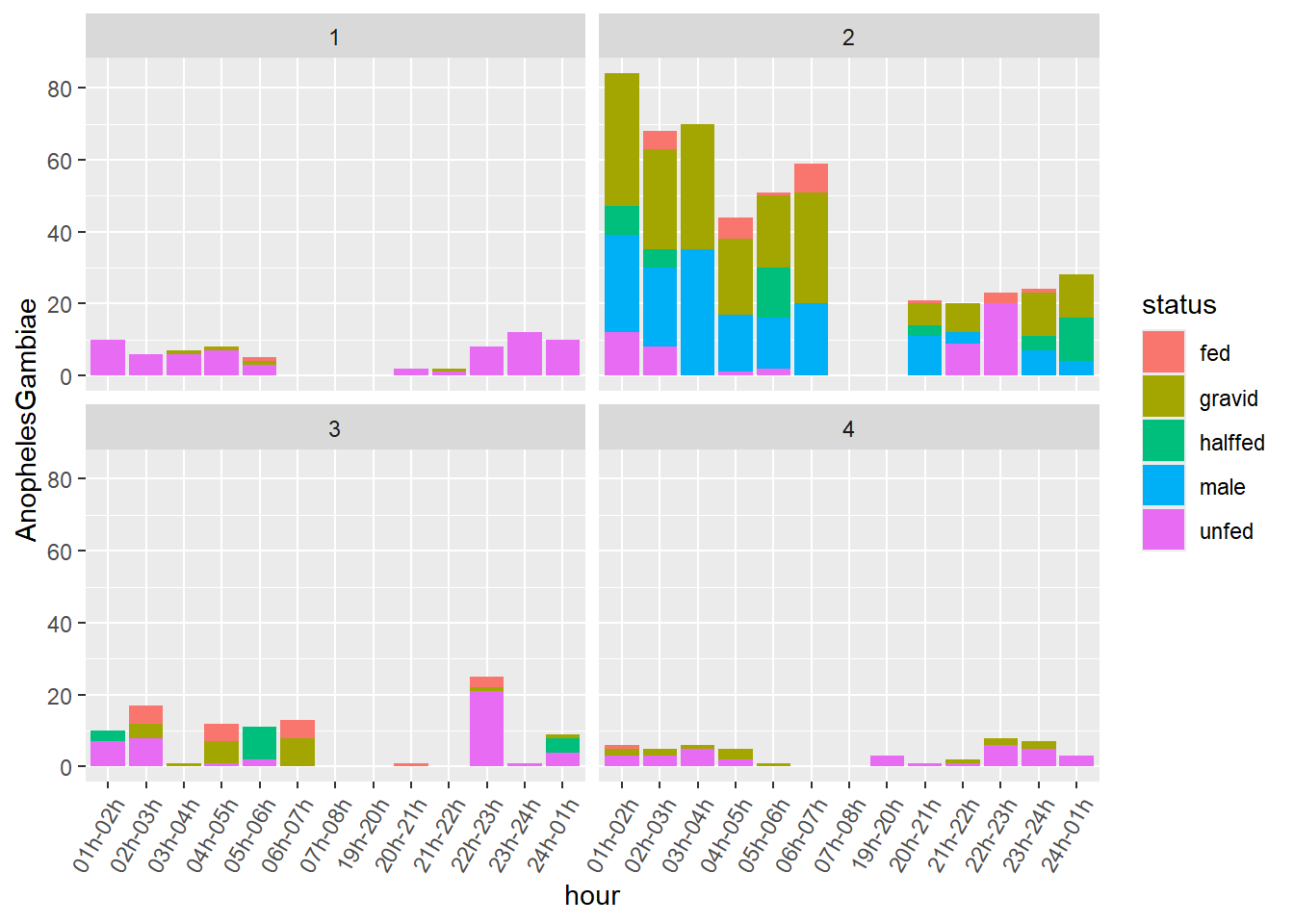
Nous pouvons aussi utiliser notre variable Anopheles_total et la représenter comme un grapique ligne au dessus du grapique en barres:
mosq_data_gamb_wide%>%
mutate(grouping=paste0(Compound.ID,Location,session))%>%
ggplot()+
geom_bar(aes(x=hour,y=AnophelesGambiae,fill=status),position="stack",stat="identity")+
geom_line(aes(x=hour,y=AnophelesGambiae_total,group=grouping))+
scale_x_discrete(guide = guide_axis(angle = 60))+
facet_wrap(~Compound.ID+session+Location)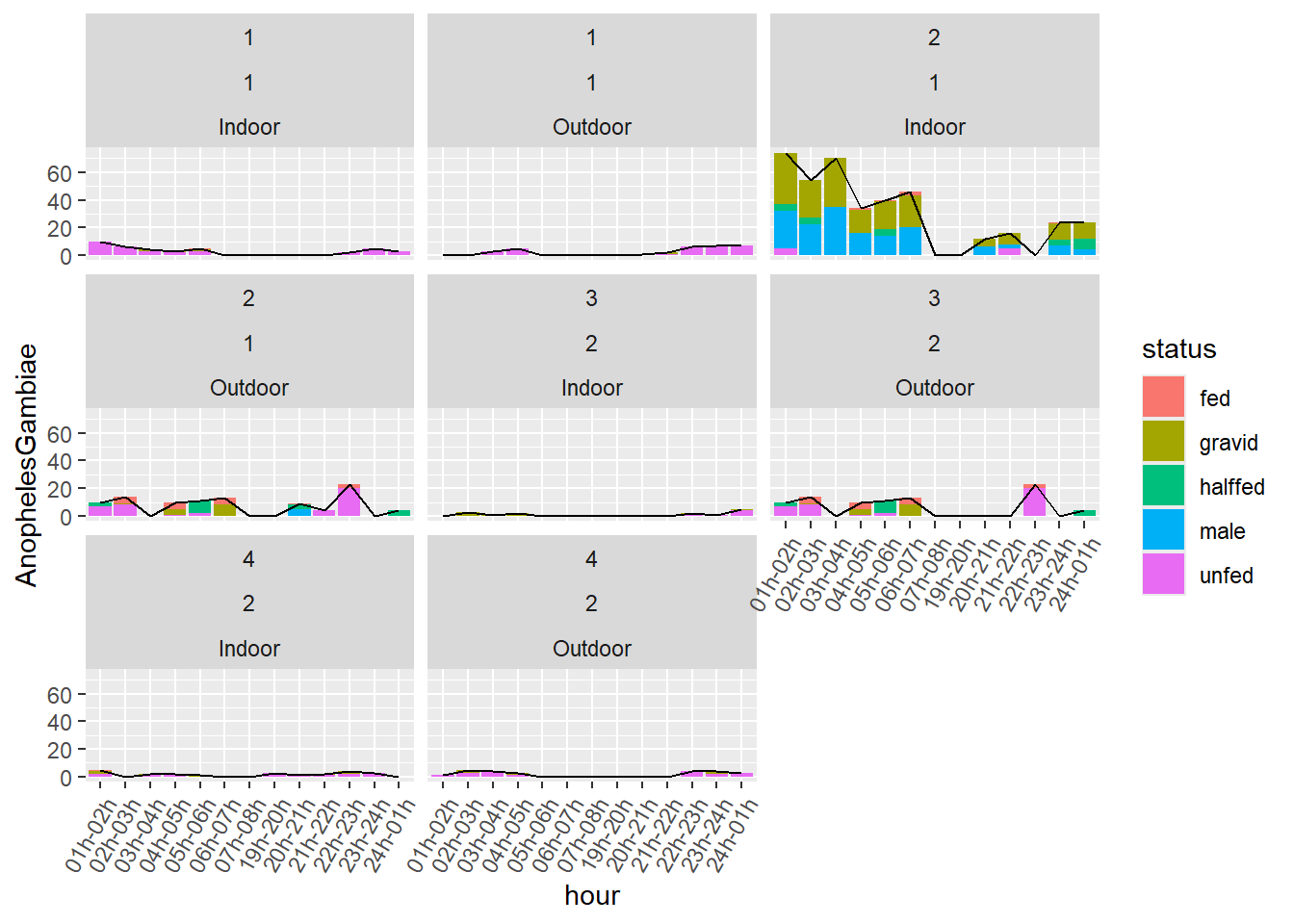
Comment procéder ?
Comparons les deux